Un moteur de recherche de taxons avec PostgreSQL, Flask et Angular
Théo Lechémia - Parc national des Ecrins / Janvier 2019
Dans le monde naturaliste, on passe beaucoup de temps à saisir des données observées sur le terrain. Que ce soit sur mobile ou sur le web, une étape importante de la saisie consiste à définir quelle espèce on a observée. L’utilisateur va potentiellement répéter cette opération une dizaine ou une vingtaine de fois. Cette opération doit donc être rapide... ce qui peut être un défi lorsqu’on recherche sur une liste de plusieurs milliers d'espèces.
Pour son confort l’utilisateur va également vouloir chercher un taxon par son nom latin, son nom vernaculaire, ou même par le synonyme sous lequel il connaît le taxon. Certains utilisateurs vont chercher plutôt par « genre » quand d’autres vont directement saisir le nom de l’espèce. La saisie se doit d’être souple et s’adapter à l’utilisateur.
Elle doit enfin être pertinente et remonter des taxons qui ressemblent aux premières lettres que l’utilisateur a tapé : celui-ci ne doit pas attendre d’avoir tapé le nom complet du taxon pour avoir des résultats pertinents.
En informatique on appelle cela de la « recherche floue » ou « fuzzy search » en anglais. C’est une méthode qui consiste à rechercher des chaînes de caractère (des enregistrements dans une base de données dans notre cas), à partir d’un « motif » approximatif, plutôt qu’à une correspondance exacte (Wikipedia).
Cette article explique comment nous avons mis en place une recherche de taxon dites de « recherche floue » dans l’application web GeoNature grâce aux données présentes dans le référentiel Taxref en construisant une Web API.
Tester la démo de ce moteur de recherche.
1. La source de données
En France, grâce au travail du MNHN, on dispose d’une base de données de tous les taxons présents sur le territoire français : le Taxref. Cette base uniformisée et partagée par tous les acteurs de acteurs de la biodiversité en France (ou presque) compte actuellement 550 843 noms de taxon. Chaque taxon dispose d’un code unique : le cd_nom et d'une série d’informations le caractérisant tel que son nom scientifique, son nom valide, sa hiérarchie taxonomique, ses synonymes etc. Le Taxref est téléchargeable ici .
Avant d’aller plus loin, petit topo sur la nature et la structuration des données dans Taxref.
- Un taxon peut posséder plusieurs noms, suivant son appellation dans différentes régions, suivant l’évolution de la taxonomie etc. On appelle les différents nom d’un taxon des synonymes.
- Chaque synonyme latin est un nouvel enregistrement dans la table Taxref.
- Les synonymes vernaculaires, c’est-à-dire les différents noms vernaculaire d’une même espèce, ne constituent pas un nouvel enregistrement. On concatène les différents noms dans la colonne nom vernaculaire séparé par des virgules.
- Le nom de « référence » pour la communauté scientifique est appelé le nom valide, ce nom valide est un nom latin suivit du nom de l’auteur (le descripteur ).
- Dans Taxref on retrouve les noms de référence grâce au code « cd_ref ». Les taxons de référence on un cd_nom égal au cd_ref.
2. Intégrer et interroger ses données sur PostgreSQL
Structurer ses données
Au Parc national des Ecrins et dans GeoNature, nous utilisons le SGBD open source PostgreSQL.
La 1ere étape est donc d’intégrer Taxref dans PostgresSQL.
Après avoir téléchargé les données, utilisez la commande « COPY » (nécessite d’être super-utilisateur PostgreSQL) pour les intégrer dans votre base :
COPY taxonomie.taxref (regne, phylum, classe, ordre, famille, sous_famille, tribu, group1_inpn,
group2_inpn, cd_nom, cd_taxsup, cd_sup, cd_ref, rang, lb_nom,
lb_auteur, nom_complet, nom_complet_html, nom_valide, nom_vern,
nom_vern_eng, habitat, fr, gf, mar, gua, sm, sb, spm, may, epa,
reu, sa, ta, taaf, pf, nc, wf, cli, url)
FROM '/tmp/TAXREFv11.txt'
WITH CSV HEADER
DELIMITER E'\t' encoding 'UTF-8';
Notre objectif est que l’utilisateur puisse saisir sa recherche autant sur le nom latin que sur le nom vernaculaire.
Or dans Taxref ces deux informations sont dans des champs différents ("lb_nom" et "nom_vernaculaire").
Nous allons donc structurer nos données pour que ces informations soit regroupées dans un seul champ, en créant une « vue matéralisée ».
La vue va contenir un champ « search_name » dans lequel on va retrouver la structure suivante :
nom_latin = nom_valide Union nom_vernaculaire = nom_valideNB : On rajoute à chaque fois le nom valide pour l'identification rapide des synonymes.
Voici la requête de création de la vue matérialisée (pour la 2ème partie de la requête, on ne met que les noms de référence puisqu’on a vu précédemment que la synonymie n’apparaît que sur le nom latin) :
SELECT cd_ref, cd_nom, CONCAT(lb_nom, ' = ', nom_valide) AS search_name FROM taxonomie.taxref UNION SELECT cd_ref, cd_nom, lb_nom, CONCAT(t3.nom_vern, ' = ', t3.nom_valide) AS search_name FROM taxonomie.taxref WHERE cd_nom=cd_ref )
Explorer et tester ses données
On peut maintenant commencer à faire des recherches dans notre vue.
En SQL on utilise le caractère ‘ %’ comme ‘wildcard’, pour rechercher n’importe quel enregistrement qui commence ou termine par une suite de caractères.
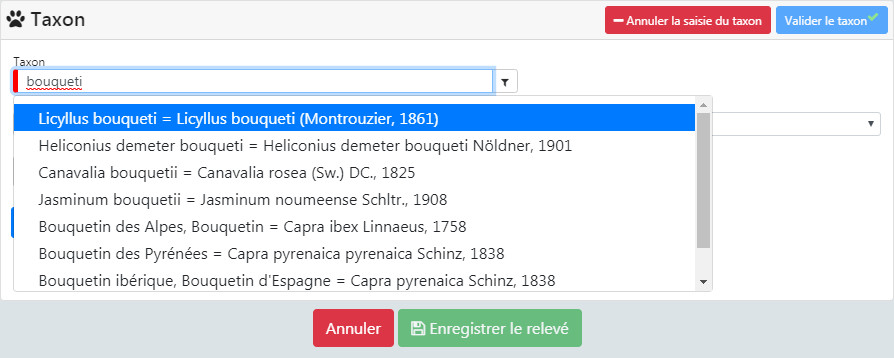
Voici par une requête qui va rechercher tous les noms qui commencent par « bouqu »
SELECT * FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE ‘bouqu % ‘
La recherche semble bien fonctionner. J’ai bien une série de noms qui commencent par « bouqu ». Le taxon auquel je pensais, le « bouquetin des alpes » arrive en 12ème position.
Je vais ensuite rechercher le même taxon sur son nom scientifique :
SELECT search_name FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE 'capra%' LIMIT 100
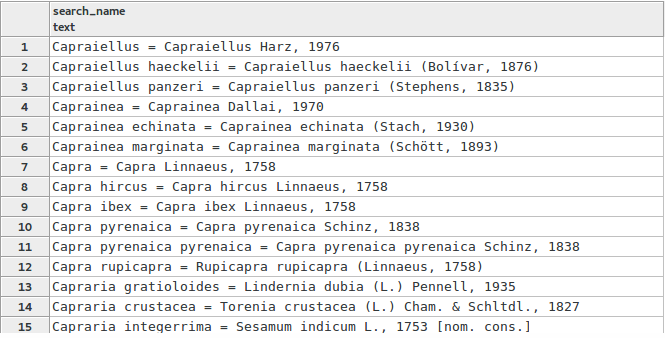
Là encore le résultat semble pertinent. Je n’ai pas changé la colonne que j’interrogeais, et j’ai bien une série de taxons dont le nom latin commence par « capra ». Notre taxon apparaît cette fois en 9ème position.
Je vais ensuite compliquer un peu les choses en recherchant seulement sur le nom de l’espèce et non sur le genre comme je viens de le faire avec « capra ». Je vais rechercher « ibex » et je vais donc devoir ajouter une wildcard pour ne plus rechercher seulement sur le début du mot, mais sur n’importe quelle série de caractères qui contient « ibex », peu importe sa position dans la chaîne de caractère.
SELECT search_name FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE ' %ibex%' LIMIT 100
Le résultat est plutôt satifsant, mais peut être déroutant. J’ai bien plusieurs taxons donc le nom bi-nominal contient « ibex », mon taxon recherché est d’ailleurs présent en 14ème et 15ème position. J’ai cependant toute une série de taxons qui ne commencent pas par « ibex », et ce peut être déstabilisant d’avoir des résultats dont le 40ème caractère contient les caractère « ibex » remonter avant le taxon que je recherchais.
Les choses se compliquent encore quand je recherche sur le nom vernaculaire avec des wildcards présentes en début et fin de mot :
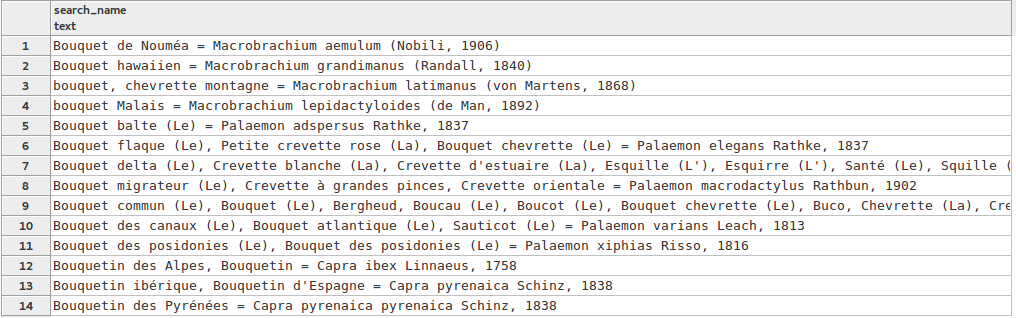
SELECT search_name FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE '%bouqu%' LIMIT 100
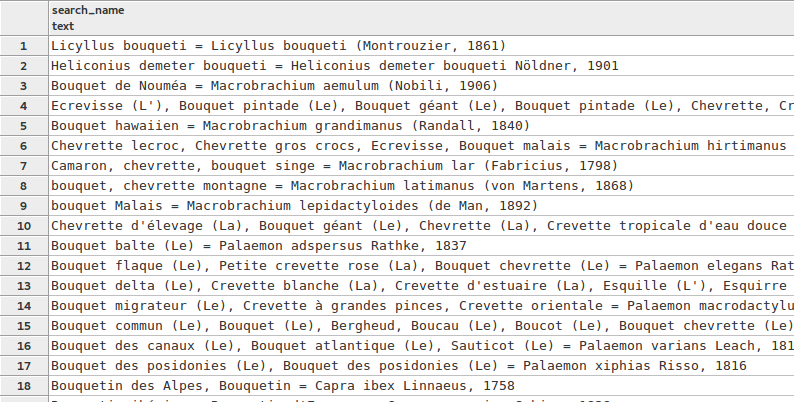
J’ai des résultats qui contiennent certes les caractères « bouqu », mais on est loin d’avoir des résultats pertinents. J’ai par exemple, ligne 6, un taxon dont le 4ème nom vernaculaire contient « bouqu », et il s’agit d’une écrevisse... Mon taxon n’apparaît lui qu’en 18ème position...
Il est donc nécessaire d’introduire un peu « d’intelligence » si on veut chercher à la fois sur le nom du genre et le nom de l’espèce.
PostgreSQL possèdent quelques extensions qui permettent de faire la « recherche floue », notamment l’extension « pg_trgm » (https://www.postgresql.org/docs/9.1/pgtrgm.html), basée sur l’algorithme du « trigram » (https://ii.nlm.nih.gov/MTI/Details/trigram.shtml). C’est un algorithme de comparaison de chaîne de caractère permettant de définir une probabilité de ressemblence entre deux chaîne de caractère. Il consiste à découper un mot ou une chaîne de caractère en bloc de trois lettres, nommé trigramme, puis de comparer ces trigrammes entre eux afin de définir des ressemblances.
L’extension permet de construire des index spéciaux pour améliorer la recherche par trigramme, et contient un certains nombre de fonction pour comparer deux chaînes de caractère. Ajoutez donc l'extension à votre base de données :
CREATE EXTENSION IF NOT EXISTS pg_trgm
La 1ère étape consiste à indexer le champs que l’on souhaite rechercher, afin que les perfomances ne soient pas trop dégradées.
CREATE INDEX trgm_idx ON taxonomie.vm_taxref_list_forautocomplete USING GIST (search_name gist_trgm_ops);
Puis on va utiliser la fonction « similarity(text,text) » qui renvoie un indice de 0 à 1 en fonction de la ressemblance de nos chaînes de caractère.
On reprend donc notre exemple précédent, avec les deux wildcards, en utilisant la fonction « similarity » et en ordonnant le résultat sur l’indice renvoyé par la fonction pour gagner en pertinence.
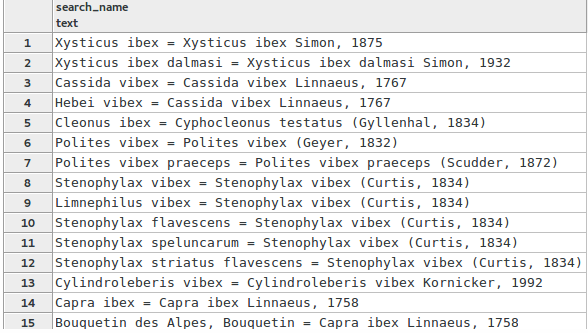
SELECT search_name, similarity(search_name, 'ibex') AS trg_incide FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE '%ibex%' ORDER BY tgr_indice DESC, search_name LIMIT 100
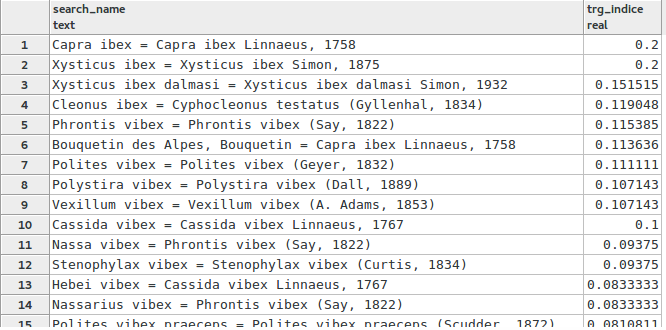
Magie ! Le bouquetin arrive en 1er. C’est dû à la répétition du mot « ibex » dans le nom latin et le nom valide.
Mais c’est plutôt positif puisque tous les taxons de référence ont par définition leur nom scientifique égal à leur nom valide !
On a donc une recherche qui va naturellement nous faire remonter les noms de référence lorsqu'on recherche sur le nom latin.
Testons sur le nom vernaculaire :
SELECT search_name, similarity(search_name, 'Bouqu') AS trg_indice FROM taxonomie.vm_taxref_list_forautocomplete WHERE search_name ILIKE '%bouqu%' ORDER BY trg_indice DESC, search_name LIMIT 100
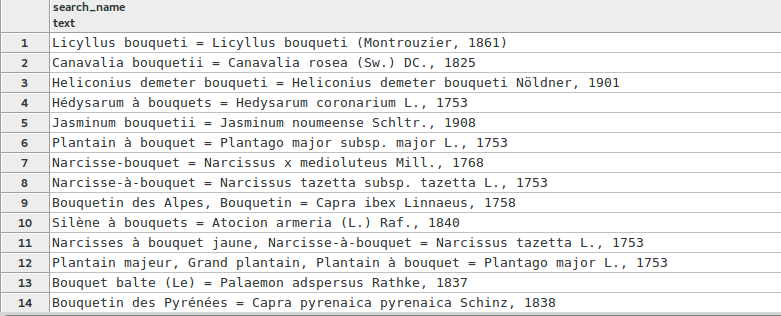
Le résultat est bien plus pertinent. Encore une fois en 1er, on a le cas d’un taxon de référence qui contient deux fois les caractères « bouqueti ». Notre recherche arrive en 9eme position, c’est mieux que le 1er essai avec une seule wildcard à la fin du mot.
3. Construire une API grâce à Flask
Flask est un microframework python (http://flask.pocoo.org/) permettant de construire rapidement des applications web et des API.
Ici on va servir de Flask pour créer une WEB-API, c’est à dire mettre à disposition URL capable d’aller intérogger notre base de données et de renvoyer des résultats sous la forme de ce que l’on vient de faire en SQL. Cette API pourra être interrogée en AJAX depuis une application web ou mobile.
Installer Flask et psycopg2 pour la connection à la BDD
pip install flask psycopg2
Voici maintenant le code permettant de créer cette petite WEB-API
# import des librairies
from flask import Flask, request
import psycopg2
from flask import Flask, request, jsonify
# création de l'application flask
app = Flask(__name__)
# ecritutre d'une fonction créeant la route de notre API
@app.route("/fuzzy_search")
def fuzzy_search():
# connection à la base
conn = psycopg2.connect(
dbname=, user=mon_user, password=
)
cur = conn.cursor()
# récupération de la query string
params = request.args
# ecriture de la requêtes SQL
query = '''
SELECT search_name, similarity(search_name,%(user_search)s AS trg_indice
FROM taxonomie.vm_taxref_list_forautocomplete
WHERE search_name ILIKE %(user_search)s
ORDER BY trg_indice DESC, search_name
LIMIT 100
'''
# on execute la requête en passant en paramètre la query_string
cur.execute(query, {'user_search' : params['q']})
# on récupère les résultats
results = cur.fetchall()
# et on ferme la connexion
cur.close()
conn.clos()
# on retourne un JSON des résultats
return jsonify(
[{'search_name': r[0], 'indice': r[1] } for r in results]
)
# Lancer le server web sur le port 5000
if __name__ == ‘main’ :
app.run(port=5000)
On a donc créé une route (une URL) qui va renvoyer les résultats de la recherche de taxons à cette adresse http://127.0.0.1:5000/fuzzy_search?q=capra.
Lorsqu’on tape l’URL dans un navigateur on a bien ce résultat :
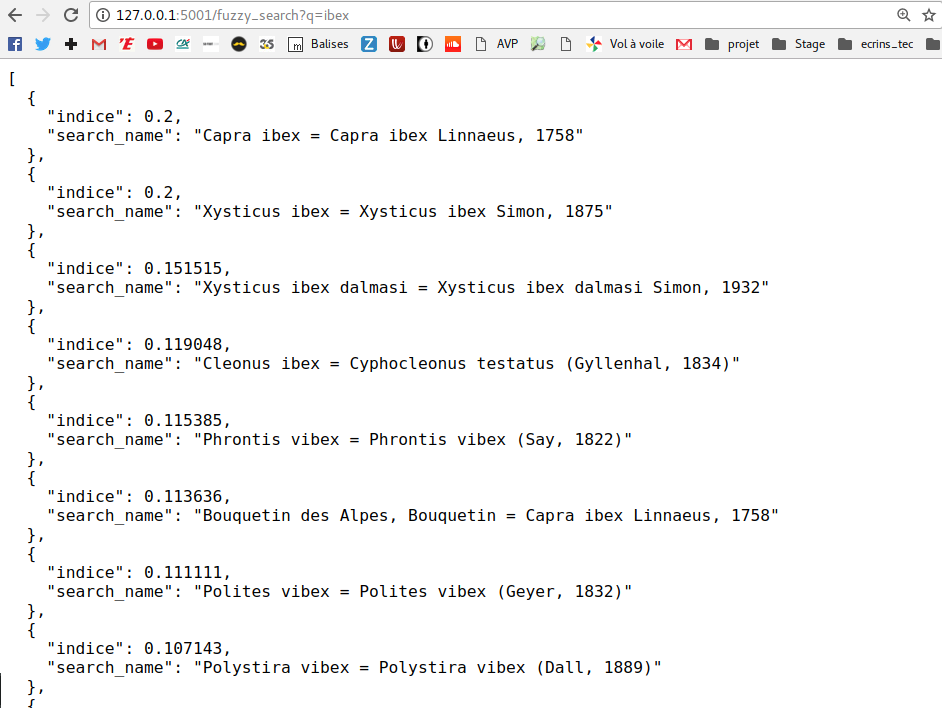
On donc a désormais une web api qui ne demande plus qu’à être interrogée depuis une page web, une application mobile ou même votre dernier logiciel de gestion de la paye !
4. Créer un composant typeahead avec Angular
On va maintenant interfacer l'API avec un petit composant Angular composé d’un simple champ de saisi où l’utilisateur va rentrer les 1ere lettres du taxon qu’il cherche, et d’une liste déroulante comprenant les résultats de l’API. Ceci est couramment appelé un « typeahead ».
Pour cela on va créer une simple balise input que l'on va connecter à un Reactive Form Angular.
HTML :<input type="text" class="form-control form-control-sm " id="taxonInput" [formControl]="taxonControl" [ngbTypeahead]="search" placeholder="Nom latin ou vernaculaire" />TS :
import { Component } from "@angular/core";
import { FormControl } from "@angular/forms";
import { HttpClient, HttpParams } from "@angular/common/http";
@Component({
selector: "app-root",
templateUrl: "./app.component.html",
})
export class AppComponent {
// On crée un formControl
public taxonControl = new FormControl();
constructor(private _http: HttpClient) {}
}
Les formulaires Angular sont basés sur le principe de programmation réactive.
La programmation réactive est un paradigme de programmation basé sur les flux de données et leur propagation dans une application.
Elle est très utilisée pour les flux de données asynhcrones et se base sur la librairie RxJS.
Dans notre cas, les évenements renvoyés par les actions de l'utilisateurs (clic sur l'input, remplissage du champ) sont considérés comme des flux de données (Observable) auxquels on va pouvoir s'abonner (Subscribe). La puissance de la librairie RxJS est qu'elle permet de contrôler ces flux de données grâce à des opérateurs. On va effet pouvoir filtrer, transformer ces évenements à notre guise.
Je vais donc dans un premier temps m'abonner à l'evenement "valueChange" de mon FormControl, c'est à dire écouter à chaque fois que l'utilisateur tape une lettre dans le champ input, puis lancer une requête HTTP à mon API en lui passant le "pattern" que l'utilisateur vient de taper. Jusque ici rien d'exceptionnel.
ngOnInit() {
this.taxonControl.valueChange.subscribe(text => {
this.getTaxons.subscribe(data => {
console.log(data);
} )
})
}
getTaxons(pattern) {
return this._http.get(
"http://demo.geonature.fr/taxhub/api/taxref/allnamebylist/100",
{ params: new HttpParams().set("search_name", pattern) }
);
Si on observe maintenant le debugger du navigateur, on voit que 7 appels au serveur été lancés après que j'ai tapé "Capra i". Chaque lettre tapée a entrainé un appel au serveur, alors qu'un seul, à la fin de la saisie utilisateur aurait suffit.
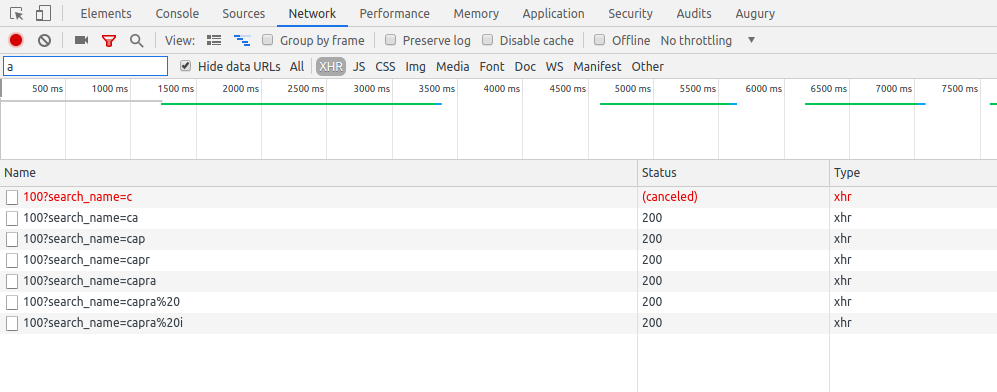
C'est ici que les opérateurs RxJS vont nous aider:
this.taxonControl.valueChange
.pipe(
filter(text => text.length >=3)
debounceTime(300)
distinctUntilChanged()
).subscribe(text => {
this.getTaxons.subscribe(data => {
console.log(data);
})
})
On va rajouter une série d'opérateur pour interferer sur le flux de données grâce à la fonction "pipe".
Le premier opérateur "filter" permet, comme sont nom l'indique, de filtrer le flux par une condition.
Si l'expression proposée est vrai, le flux passe au prochain opérateur sinon il est arreté.
On filtre donc les chaines superieures ou égales à 3 caractères pour éviter de chercher sur les premiers charactères qui sont peut discriminants.
Le second opérateur ajoute un "temps de latence" au flux, ici l'evennement renvoyé à chaque lettre tapée est retardé de 300 millisecondes
(seul le dernier évenement passe alors le flux). Enfin le dernier opérateur permet de ne pas lancer deux fois la même recherche
(Par exemple si je tape "capra ib" puis "capra ibex" et que je reviens a "capra ib", les deux dernières requêtes ne seront pas lancées).
Il existe une toute une série d'autres opérateurs qui peuvent être utilisés dans d'autres contextes.
On utilise finalement un composant bootstrap typeahead
pour rendre l'interface utilisateur plus ergonomique.
Ce composant attend directement un "Observable", on doit donc légerement modifier le code :
search = (text$: Observable) => text$.pipe( debounceTime(300), distinctUntilChanged() switchMap(term => this.getTaxons(term).pipe( catchError(() => { return of([]); })) )) )
Grâce à l'utilisation des concepts de programmation réactive (flux de données et opérateurs) on obtient une recherche plus "intelligente" qui soulage très largement le serveur de requêtes inutiles.
Tester la démo ici.